au sommaire
- 1.6.1. ISOLEMENT DES GRANULOCYTES NEUTROPHILES
- 1.6.1.1. Principe
- 1.6.1.2. Protocole
- 1.6.2. EVALUATION DE LA CYTOTOXICITE DE LA MOLECULE A TESTER
- 1.6.2. 1. Principe
- 1.6.2. 2. Protocole
- 1.6.3. DOSAGE DE L'ANION SUPEROXYDE EN MODELE ACELLULAIRE
- 1.6.3.1. Principe
- 1.6.3.2. Protocole
- 1.6.4. DOSAGE DE L'ANION SUPEROXYDE EN MODELE CELLULAIRE
- 1.6.4.1. Principe
- 1.6.4.2. Protocole
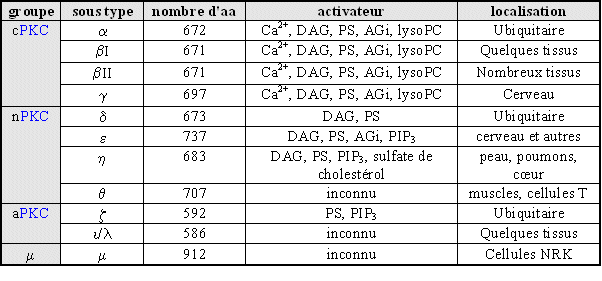
- 1.6.5. PURIFICATION DE LA PKC PAR CHROMATOGRAPHIE
- 1.6.5.1. Principe
- 1.6.5.2. Protocole
- 1.6.6. ETUDE DE LIAISON
- 1.6.6.1. Principe
- 1.6.6.2. Protocole
- 1.6.7. WESTERN BLOT
- 1.6.7.1. Principe
- 1.6.7.2. Protocole
- 1.6.8. ETUDES STATISTIQUES
- À lire aussi
Les manipulations décrites sont réalisées à 4°C, sauf indication contraire.
1.6.1. ISOLEMENT DES GRANULOCYTES NEUTROPHILES
1.6.1.1. Principe
Les granulocytesgranulocytes neutrophilesneutrophiles sont séparés des lymphocyteslymphocytes et des monocytesmonocytes par un gradient de densité obtenu par une solution d'Histopaque-1077® (Sigma). Le culot composé de globules rougesglobules rouges et de granulocytes neutrophiles est remis en suspension dans une solution de chlorure d'ammonium afin d'effectuer une hémolysehémolyse. Les cellules sont recueillies et lavées après centrifugation.
1.6.1.2. Protocole
Les granulocytes neutrophiles sont prélevés sur héparinate de lithium à partir de sang de volontaires sains. Le sang est dilué au demi dans un tampon Na2HPO4 (PBS) à 0,1 M et à pH 7,4. Au fond de chaque tube, 10 ml d'Histopaque-1077 (Sigma) sont déposés. Les hématies et les granulocytes neutrophiles sont séparés des autres cellules sanguines après une centrifugation à 400g pendant 30 minutes à température ambiante. Ensuite, 10 ml de plasma sont prélevés et mis de côté, le reste du surnageant est éliminé afin de ne conserver que le culot globulaireglobulaire. Les cellules sont remises en suspension dans les 10 ml de plasma, et diluées au 1/10 grâce à une solution isotonique de chlorure d'ammonium (cf. annexe 1), l'hémolyse dure 12 minutes. Les tubes sont ensuite centrifugés à 400 g pendant 10 minutes à 4°C, le surnageant est éliminé et les culots lavés par une solution de Hanks'HEPES (cf. annexe 1). Les cellules sont remises en suspension dans la même solution puis centrifugées dans les mêmes conditions que précédemment. Enfin, le surnageant est prélevé et les cellules remises en suspension dans 20 ml de Hanks'HEPES et conservées à 4°C (Cabanis A. et al., 1994).
Une numération est faite sur cellule de Thoma. La vitalité des granulocytes neutrophiles est évaluée par un bleu de Trypan. Pour cela, 0,3ml de la suspension sont mélangés avec 0,1ml d'une solution native de bleu Trypan et mis à incuber à 37°C pendant 10 minutes. Le pourcentage de cellules ne prenant pas la coloration du bleu de Trypan correspond au pourcentage de vitalité cellulaire. L'isolement des granulocytes neutrophiles est ainsi réalisé avec un degré de pureté de 95%, la viabilité est également de 95%.
1.6.2. EVALUATION DE LA CYTOTOXICITE DE LA MOLECULE A TESTER
1.6.2. 1. Principe
La lactatelactate déshydrogénase est une enzyme exclusivement cytoplasmique et relativement stable. L'augmentation de l'activité de cette enzyme dans le surnageant des cellules permet de détecter une altération de la perméabilité membranaire.
Le dosagedosage est fondé sur la réaction suivante :
PyruvatePyruvate + NADH,H+ ---> L-lactate + NAD+
Il consiste en la détermination cinétique de l'activité de la lactate déshydrogénase avec suivi de l'absorbance du NADH à la longueur d'onde de 340 nm.
1.6.2. 2. Protocole
La cytotoxicité de la molécule à tester est évaluée par le dosage de l'activité enzymatiqueenzymatique de la lactate déshydrogénase (LDH) du surnageant cellulaire d'une suspension de granulocytes neutrophiles (5.106 GrN/ml) incubée à 37°C avec l'inhibiteur à différentes concentrations. Les tubes sont centrifugés 5 minutes à 700 g, les surnageants sont placés dans les cuves spectrophotométriques. L'activité est mesurée à l'aide de NADH 0,2 mM et de pyruvate 1,6 mM; la cinétique est réalisée à 30°C et lue à 340 nm. L'activité maximale de la LDH libérée dans le surnageant est dosée après lyse des cellules par ajout d'eau distilléeeau distillée (Levert H. et al., 1998).
Le pourcentage de lactate déshydrogénase relarguée est calculé à partir de l'activité maximale de cette enzyme :
activité LDH totale
X=(activité LDH surnageant de référence)+(activité LDH du lysat)
activité LDH avec inhibiteur (I)
Y=(activité LDH du surnageant avec I)-(activité LDH du surnageant de référence)
%activité LDH=(Y/X)x100
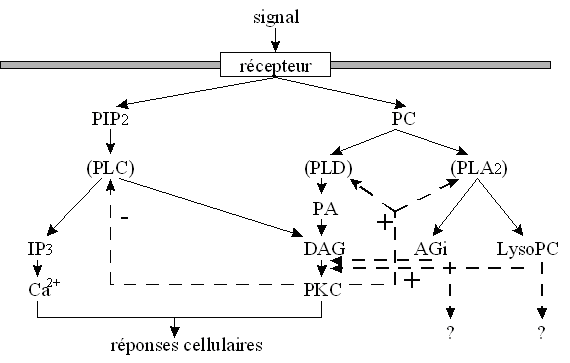
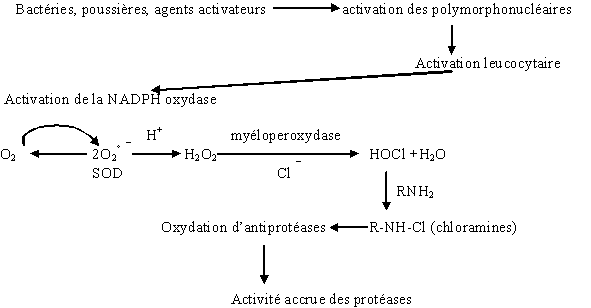
1.6.3. DOSAGE DE L'ANION SUPEROXYDE EN MODELE ACELLULAIRE
1.6.3.1. Principe
L'anionanion superoxyde est produit par le système enzymatique xanthine-xanthine oxydaseoxydase. La quantité produite par ce système, identique à celle libérée par les granulocytes neutrophiles, est mesurée par réduction du ferricytochrome C. Cette réduction s'accompagne d'un changement de coloration détecté par l'absorbance à 550 nm. La concentration en anion superoxyde est calculée à partir du coefficient d'extinctionextinction molairemolaire du ferricytochrome C (E550nm =2,1.10-2 µM-1.cm-1).
1.6.3.2. Protocole
Le milieu réactionnel est composé d'EDTA 1 mM, de xanthine 1 mM, de ferricytochrome C 20 µM et de différentes concentrations en molécules à tester. La production de l'anion superoxyde est déclenchée par ajout de xanthine oxydase à 0,1 U.I./ml. L'absorbance est mesurée à 550nm contre des tubes ne contenant ni xanthine, ni xanthine oxydase (Khalfi F. et al., 1996).
1.6.4. DOSAGE DE L'ANION SUPEROXYDE EN MODELE CELLULAIRE
1.6.4.1. Principe
La quantité d'anions superoxyde est mesurée en utilisant la réduction du ferricytochrome C (FC) par cet anion. Cette réaction s'accompagne d'un changement de coloration du ferricytochrome C détecté à 550 nm au spectrophotomètre. La concentration de l'anion est calculée grâce au cœfficient d'extinction molaire du FC réduit : E550 nm=2,1.10-2 µM-1.cm-1 (Cohen H.J. et Chovaniec M.E., 1978).
1.6.4.2. Protocole
Les granulocytes neutrophiles (106 cellules à la concentration de 106 cellules/ml) sont pré-incubés avec l'inhibiteur potentiel à 37°C pendant 15 minutes. Puis les cellules sont activées soit par le fMLP 1 µM soit par le PMAPMA 160 nM à 37°C pendant 15 minutes. Une fois cette première incubation réalisée, le ferricytochrome C à 0,2 mg/ml est ajouté au milieu, les tubes sont alors laissés au bain-marie pendant 10 minutes. Enfin, les différents milieux sont centrifugés à 700g pendant 5 minutes à 4°C afin d'éliminer les cellules. Les surnageants sont mis dans des cuves spectrophotométriques, l'absorbance est mesurée à 550 nm contre des surnageants des référence (de même composition que précédemment), auxquels il a été ajouté, au départ, de la superoxyde dismutase à 210 U.I./ml; cette dernière accélère la dismutationdismutation de l'anion superoxyde en peroxyde d'hydrogèneperoxyde d'hydrogène empêchant la réduction du ferricytochrome C (Cabanis A. et al., 1996).
1.6.5. PURIFICATION DE LA PKC PAR CHROMATOGRAPHIE
1.6.5.1. Principe
Afin de limiter au maximum les liaisons non spécifiques lors de l'étude de liaison, nous avons pris la décision de purifier l'extrait cellulaire servant à ces expérimentations. Pour cela les granulocytes neutrophiles sont lysés puis mis en contact avec le gelgel servant à la chromatographiechromatographie. L'ensemble des protéinesprotéines est capable de s'adsorber aux billes de DEAE-Sephacel par des liaisons faibles. L'ensemble est ensuite lavé afin d'éliminer une première partie des composants contenus dans l'extrait cellulaire, l'élution des protéines kinaseskinases C se fait en additionnant au tampon contenant du chlorure de sodiumsodium afin d'augmenter la force ionique. Ceci a pour conséquence de décrocher les isoenzymes de la phase stationnaire et donc de les récupérer partiellement purifiées dans la phase mobilemobile, sachant qu'une dernière partie des constituants se trouve toujours à l'intérieur de la colonne de chromatographie.
1.6.5.2. Protocole
Afin d'éviter une dégradation de la protéine kinase C par des sérinesérine-thréoninethréonine protéasesprotéases, les granulocytes neutrophiles, à la concentration de 5.106 cellules/ml de Hanks'HEPES, sont incubés deux fois 15 minutes avec une solution de fluorure de phénylméthylsulfonyl (PMSF) à 57 µM. Après une centrifugation à 400 g pendant 10 minutes à 4°C, les granulocytes sont remis en suspension dans le tampon de lyse de chromatographie (cf. annexe 1) à la concentration de 50.106 cellules/ml et à raison de 25.106 cellules par tube. Les cellules sont lysées par sonication, à raison 6 fois 5 secondes en prenant garde à bien refroidir les tubes entre chacune des phases de sonication pour éviter une dénaturationdénaturation trop importante de l'enzyme. Le lysat obtenu est centrifugé à 100 000g pendant 30 minutes à 4°C, le surnageant est récupéré. Il est ensuite mélangé à volumevolume / volume avec du DEAE-Sephacel (Pharmacia Biotech). Le tout est mis en légère agitation pendant une heure, la matrice est mise dans la colonne puis lavée par 10ml du tampon de lyse. L'élution de la protéine kinase C est obtenue par addition de chlorure de sodium pour arriver à la concentration de 150 mM. L'élution est pratiquée de manière isocratique (Nakanishi H. et Exton J., 1992).
Les échantillons récupérés peuvent être conservés à -80°C pendant une semaine, à condition d'ajouter au milieu du PMSF à la concentration de 57 µM.
La mise en évidence de la présence de la PKC dans chacune des fractions recueillies est faite par western blotwestern blot ( technique décrite plus loin).
Pour être utilisée dans l'étude de liaison ou dans la mesure d'activité, il est nécessaire de quantifier la protéine kinase C, ceci est fait par un dosage de Lowry. Pour cela, une gamme étalon est réalisée en utilisant une solution de sérumsérum albuminealbumine comme source de protéines, chaque tube de cette gamme et chaque tube échantillon à doser faisant 0,5 ml. Pour chacun, 2,5 ml de réactifsréactifs A (cf. annexe 1) sont ajoutés, les tubes sont mélangés soigneusement et incubés pendant 10 minutes à température ambiante. La coloration est alors développée par addition de 0,25 ml de réactif de Folin précédemment dilué au demi, les tubes sont à nouveau mis en incubation pendant 30 minutes à température ambiante. La lecture de l'absorbance se fait à 750 nm contre l'airair.
1.6.6. ETUDE DE LIAISON
1.6.6.1. Principe
Cette étude de liaison est effectuée avec un ligandligand radiomarqué : le 3H phorbol-12,13-dibutyrate (3H PDB) à 18,6 Ci/mmol.
L'extrait cellulaire partiellement purifié, est incubé en présence d'un ligand marqué qui est spécifique de la protéine kinase C. Cette molécule se fixe par des liaisons faibles sur un site précis de l'enzyme. Une fois cette étape effectuée, le milieu est filtré et lavé afin d'éliminer le ligand en solution ou fixé sur d'autres constituants et plus faiblement que sur la protéine kinase C. La radioactivitéradioactivité est mesurée sur le filtratfiltrat et par corrélation donne la quantité d'esterester de phorbol toujours présente après les lavages.
1.6.6.2. Protocole
La quantité d'enzyme utilisée est de 20 µg, elle est mise dans un tampon spécifique (cf. annexe 1), le volume final est de 325 µl. La liaison non spécifique est déterminée par ajout de phorbol-12,13-dibutyrate froid à une concentration de 30 µM.
Tout d'abord, l'enzyme est incubée à 30°C en présence de l'inhibiteur pendant 15 minutes, mais en absence du ligand radiomarqué. Ensuite, le 3H PDB à la concentration de 30 nM est additionné au milieu réactionnel afin de démarrer la réaction, les tubes sont laissés à 30 °C pour une duréedurée de 10 minutes. L'arrêt de la réaction se fait par addition de 1 ml de DMSO à 0,5% glacé. Le tout est déposé sur des filtres (GF/C Whatman) préalablement mis dans une solution de polyéthylènimine à 0,3%. Ces filtres sont lavés par 5 fois par 1 ml de DMSO puis sont séchés par aspiration. Le comptage se fait par un compteur à scintillation (Wang J.P. et al., 1995).
1.6.7. WESTERN BLOT
1.6.7.1. Principe
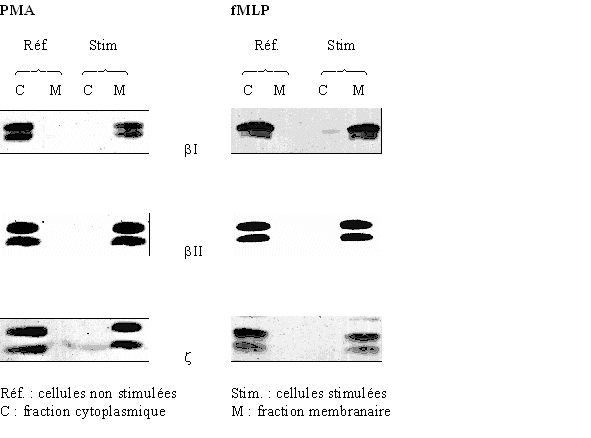
Nous utilisons la technique de western blot afin de caractériser la présence de la protéine kinase C (après chromatographie) ou son déplacement intracellulaire au cours de son activation (étude de la translocationtranslocation).
Pour la translocation, après incubation des granulocytes neutrophiles avec un stimulant, les fractions membranaires et cytosoliques sont séparées par ultracentrifugation. Ensuite, les protéines sont dénaturées à 100°C en présence de sodium dodécyl sulfate (SDS). Ceci entraîne la suppression des structures secondaires, tertiaires et quaternairesquaternaires des protéines permettant ainsi une fixation du SDS sur ces protéines et donc de les charger négativement (transformation en anions). La migration électrophorétique ne se fera donc plus qu'en fonction de la massemasse moléculaire de chaque protéine, étant donné que chacune possède maintenant la même charge. L'électrophorèseélectrophorèse a pour but de séparer les protéines sur un gel de polyacrylamide, ensuite ce gel est mis en contact avec une membrane de nitrocellulose et, un courant allant du gel vers la membrane permet de fixer les protéines contenues dans le gel sur cette membrane. Le but ensuite est de pouvoir repérer les protéines étudiées sur le support. Pour cela, la membrane est incubée avec un anticorpsanticorps primaire capable de reconnaître la protéine recherchée et de la fixer, puis un anticorps secondaire marqué à la peroxydase est ajouté au milieu afin qu'il puisse se fixer sur le premier anticorps. La membrane est alors incubée avec du luminolluminol qui est alors dégradée par la peroxydase libére de l'énergieénergie lumineuse capable d'impressionner un film photographique hypersensible. Les protéines apparaissent alors sous la forme de bandes sombres sur le film.
1.6.7.2. Protocole
Les granulocytes neutrophiles sont dans du tampon Hanks'HEPES à la concentration de 5.106 cellules/ml et à raison de 25.106 cellules par tube. Deux incubations de 15 minutes chacune sont réalisées avec du PMSF à 57 µM, ceci pour limiter la dégradation de la protéine kinase C. Ensuite, chaque milieu est mis en incubation avec l'inhibiteur et/ou l'activateur à 37°C, durant un temps donné par l'expérience, selon ce que l'on désire mettre en évidence. Dans tous les cas, l'arrêt de la stimulationstimulation se fait en ajoutant 4 volumes de Hanks'HEPES glacé. Chaque milieu est mis à centrifuger à 400 g pendant 10 minutes à 4°C afin de récupérer le culot cellulaire.
Les cellules sont remises en suspension dans le tampon de lyse pour western blot (cf. annexe 1) à la concentration de 50.106 cellules /ml. Elles sont soniquées 6 fois 5 secondes, en refroidissant les tubes entre chaque passage. Les milieux sont centrifugés à 8000 g pendant 5 minutes à 4°C pour éliminer les noyaux et les cellules non lysées. Les surnageants sont récupérés et centrifugés à 100 000g pendant 30 minutes à 4°C afin de séparer le culot contenant les membranes et le surnageant contenant la fraction cytoplasmique. La fraction membranaire est remise en suspension dans 200 µl du tampon de lyse (Hansra G. et al., 1999).
Tous les échantillons sont dilués au tiers par une solution échantillon (cf. annexe 1) et les milieux sont dénaturés pendant 10 minutes dans un bain-marie à 100°C.
L'électrophorèse, adaptée pour le système PROTEAN II (Biorad), est réalisée à 20°C sur un gel de polyacrylamide à 10 % en présence de sodium dodécyl sulfate (SDS-PAGE) (Laemmli U.K., 1970).
Dans chaque puits il est déposé l'équivalent de 130 µg de protéines soit 90 µl de la fraction membranaire et 100 µl de la fraction cytoplasmique. La migration électrophorétique se fait à 50 V et pour une durée d'environ 15 heures, en fait c'est le front de migration formé par le bleu de bromophénol qui indique le moment où l'électrophorèse doit être arrêtée.
Les protéines contenues dans le gel sont ensuite transférées sur une membrane de nitrocellulose. Pour cela le gel et la membrane sont mis en sandwiches entre deux feuilles de papier Whatman et deux tampons en fibre, puis l'ensemble est traversé par un courant électriquecourant électrique afin que les protéines se fixent sur la membrane. Cette étape se fait à 24 V pendant 3 heures et à température ambiante. La qualité du transfert est vérifiée en colorant la membrane avec une solution de rouge de Ponceau à 1%, ce qui permet de visualiser l'ensemble des protéines présentes sur la membrane de nitrocellulose.
Cette membrane est incubée pendant 1,5 heure avec une solution pour western blot (cf. annexe 1) contenant du lait à la concentration de 10 %, ceci permettra d'éviter des liaisons non spécifiques des anticorps utilisés par la suite. La solution pour western blot est ensuite employée pour laver la membrane 4 fois 10 minutes. L'anticorps primaire polyclonal de chèvre (TEBU), correspondant à l'isoforme de la protéine kinase C recherchée, est dilué 1000 fois dans la solution pour western blot à laquelle il est ajouté du lait et de la sérum albumine bovine concentrée à 1%, puis il est mis en contact avec la membrane pendant une nuit à 4°C ou pendant 1,5 heure à température ambiante. La membrane est ensuite lavée 4 fois 10 minutes avant de mettre l'anticorps secondaire de singe (Nordic), marqué à la peroxydase, et qui est dirigé contre les chaînes lourdes et légères des anticorps primaires. Il est dilué 10 000 fois dans le même tampon que l'anticorps primaire, cette étape se fait pendant 45 minutes à température ambiante. L'excès d'anticorps est éliminé par quatre lavages de 10 minutes.
Les protéines sont maintenant marquées spécifiquement et peuvent être révélées par le kit ECL (Enhanced Chemiluminescence, Amersham). La peroxydase marquant l'anticorps secondaire oxyde le luminol en présence de peroxyde d'hydrogène. Le luminol excité revient à son état initial en émettant une lumièrelumière à 428 nm détectée par un film photographique (Hyperfilm ECL, Amersham). La membrane est mise au contact du luminol pendant une minute, puis avec le film photographique pour une durée de 30 minutes. La révélation du film est arrêtée lorsque les taches sont suffisamment nettes et marquées. Ceci permet de visualiser la présence des zones sombres correspondant à l'isoforme de la protéine kinase C recherchée.
La membrane est reprise afin d'être utilisée pour détecter une autre isoenzyme. Elle est mise dans une solution de glycineglycine à 0,1 M à 50°C en pratiquant une légère agitation. Plusieurs lavages successifs sont réalisés afin d'éliminer la totalité de la glycine, la suite du protocoleprotocole est la même que précédemment, à partir de l'étape de saturation de la membrane.
1.6.8. ETUDES STATISTIQUES
Les résultats ont été soumis à une étude statistique utilisant un test non paramétrique (Student), où les valeurs p