au sommaire
- À lire aussi
Une extraordinaire diversité
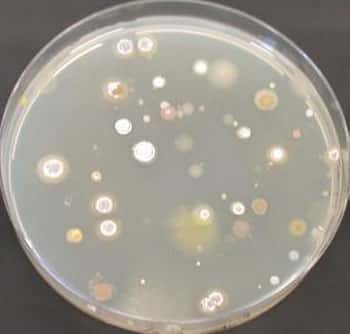
Des bactériesbactéries partout, mais qui sont-elles ? Le besoin de procéder à des études de taxonomietaxonomie existe depuis le début de l'histoire de la microbiologie, comme dans toutes les autres disciplines de la biologie. Il faut décrire et classer les espècesespèces en fonction d'un certain nombre de critères. Avant l'essor de la biologie moléculairebiologie moléculaire, les espèces étaient décrites en fonction de paramètres physiologiques (taille, forme, couleur, mobilité etc...) et biochimiques (capacité à métaboliser tel ou tel composé etc...). Cependant ces tests sont rendus imprécis par la variabilité des réponses d'une souche à l'autre ou pour une même souche, en fonction du temps et du milieu sur lequel les tests sont effectués.

Stratégie utilisée pour l'analyse moléculaire de la diversité
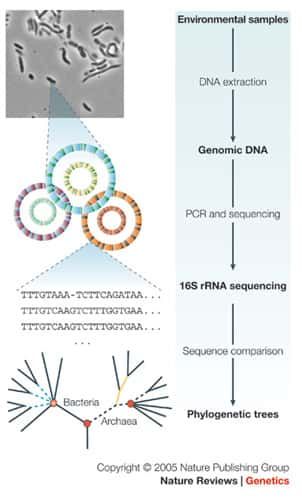
Il a fallu attendre les progrès conjoints de la biologie moléculaire (amplification en chaîne d'ADNADN, séquençageséquençage à haut-débitdébit) et de la bioinformatique pour que se développe la phylogénie moléculairephylogénie moléculaire. Il est alors devenu possible d'établir des arbresarbres généalogiques à partir des séquences d'un même gènegène comparées entre toutes les espèces. Le concept d'horloge moléculairehorloge moléculaire prédit qu'un gène donné évolue, en fonction des contraintes sélectives, par mutations successives à une vitesse constante qui lui est propre. La mesure de ces mutations reflète donc l'histoire évolutive des espèces. Carl Woese a été le premier, en 1987, à proposer l'utilisation du gène codant pour l'ARNARN ribosomique 16S, comme marqueur de phylogénie. Ce gène de 1500 paires de bases répond à plusieurs critères essentiels pour la taxonomie : (1) il est universel c'est-à-dire présent chez tous les procaryotesprocaryotes, où il remplit la même fonction (2) il évolue lentement (3) il peut être facilement séquencé car il possède des domaines hautement conservés avec peu de mutations fixées au cours de l'évolution et enfin, (4) il présente des régions de séquence plus variable qui sont d'autant plus différentes entre deux espèces que celles-ci sont phylogénétiquement éloignées. Ainsi, pour positionner dans un groupe phylogénétiquephylogénétique une nouvelle espèce, on procède à l'amplification et au séquençage de ce gène puis à sa comparaison avec les séquences déjà connues disponibles dans les banques de données.

Arbre phylogenetique représentant les principaux phylumsphylums des Bacteria: illustre la présence de groupes entiers sans représentants cultivables (sur la figure: en noir et en blanc: groupes avec représentants cultivables; en gris: sans représentants cultivables) © Rappé MS & Giovannoni SJ (2003) Ann. Rev. Microbiol. 57:369-394
La classification basée sur l'ADNr 16S a eu de profondes conséquences sur notre vision du monde bactérien. D'une part, elle est clairement différente de celle établit sur les critères physiologiques et a induit une refonte de l'arbre phylogénétiquearbre phylogénétique des bactéries cultivables. Mais, plus important encore, elle a permis de mettre en évidence des groupes phylétiques entiers n'ayant aucun représentant cultivable. En effet, des études de diversité moléculaire, affranchies de l'étape de mise en culture, ont dévoilé l'extraordinaire richesse du monde bactérien. Le nombre total d'espèces a été estimé à 1 million, alors que 5000 environ sont décrites plus ou moins bien actuellement. Un immense champ de recherche est ouvert !