au sommaire
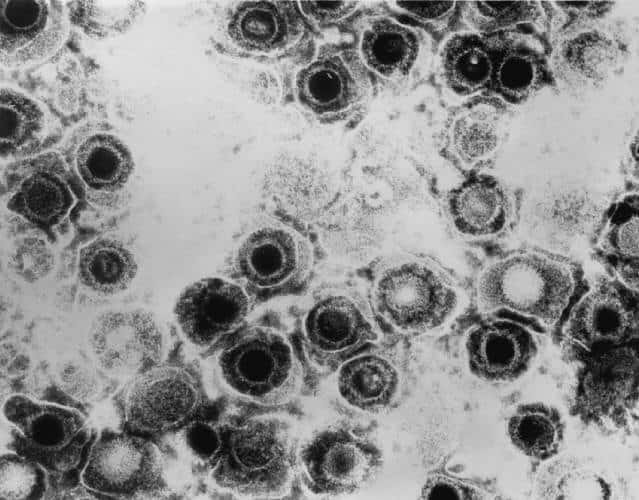
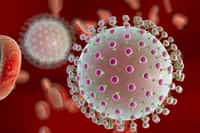
La multiplication du virus de l'herpès, vu ici au microscope électronique, peut être inhibée grâce à l'augmentation du microARN miR-199a-3p dans la cellule. Crédits DR.
Les virus sont des parasitesparasites cellulaires, c'est-à-dire qu'ils sont entièrement dépendants de la présence de cellules hôtes pour pouvoir se multiplier. Les virus possèdent leur propre génome, et codent pour des protéines capables de copier leur acide nucléique (réplicase), mais d'autres éléments nécessaires au cycle viral, disponibles dans la cellule, sont accaparés par le virus. Sans cet environnement favorable, le virus ne peut pas se multiplier.
Les antiviraux ciblent principalement les virus eux-mêmes, en inhibant leur réplicase par exemple. Cette méthode est efficace, mais peu durable dans le temps : certains virus ont la particularité de muter rapidement et de pouvoir devenir résistants. D'autres stratégies antivirales seraient donc les bienvenues : l'environnement cellulaire étant aussi nécessaire au virus, pourquoi ne pas le modifier pour le rendre inhospitalier ?
C'est ce qu'ont tenté de faire une équipe de l'Université d’Edimbourg. Ils ont mis au point une méthode, publiée dans le journal PNAS, pour tester l'effet, sur l'infection virale, de l'augmentation ou de la diminution de certaines moléculesmolécules naturellement présentes dans la cellule : les microARN.
Composés d'une vingtaine de nucléotidesnucléotides, les microARNmicroARN régulent l'expression d'un grand nombre de gènesgènes : tous les ARNARN possédant une séquence proche du microARN sont dégradés, menant à la diminution de la synthèse de la protéine correspondante. L'effet peut ensuite se répercuter sur une cascade de gènes et de protéines, modifiant l'environnement intracellulaire.
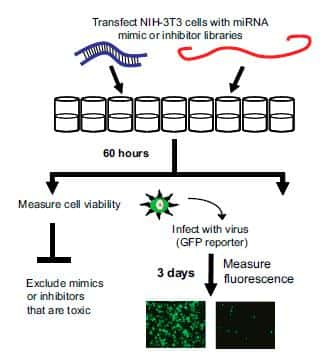
Stratégie du criblage de l'effet des microARN sur l'infection virale. Le mime (bleu) ou le piège (rouge) du microARN est inséré dans les cellules animales. Après 60 heures, la toxicité des microARN est vérifiée (à gauche), puis les cellules sont infectées par du virus fluorescent (à droite). Trois jours plus tard, la fluorescence des cellules est mesurée. © Université d'Edimbourg / PNAS
Un microARN, plusieurs virus affectés
Pour augmenter ou diminuer la quantité des microARN dans les cellules, les chercheurs ont utilisé des molécules qui les miment (un microARN identique), ou qui les piègent (un ARN complémentaire qui va séquestrer le microARN). Ces molécules (312 différentes) ont été insérées dans des cellules animales in vitroin vitro. Si les molécules ne se sont pas avérées toxiques, ces mêmes cellules ont ensuite été infectées, 60 heures plus tard, par des virus modifiés pour exprimer une protéine fluorescente (GFP, green fluorescent protein). Après trois jours de culture dans des conditions favorables à la réplicationréplication virale, les cellules ont été analysées pour déterminer la quantité de fluorescence émise, qui sera donc proportionnelle à la quantité de virus.
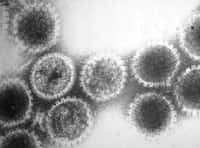
Les chercheurs ont testé plusieurs virus : le virus de l'herpèsherpès, le cytomégalovirus, le virus de la forêt de Semliki. Ces virus n'appartiennent pas à la même famille, et n'utilisent pas la même stratégie de réplication au sein de la cellule. Pourtant, alors que certaines des molécules n'ont pas d'effet sur la multiplication virale ou encore que d'autres l'améliorent, certaines molécules, à l'image de miRmiR-199a-3p, sont capables de réduire l'accumulation de l'ensemble de ces virus.
Les chercheurs n'ont pas pu déterminer si l'action de miR-199a-3p est réellement indirecte (il modifie l'environnement intra-cellulaire) ou directe (il agit sur le virus lui-même). Toutefois, la stratégie de criblage de molécule mise au point pourrait être menée à plus grande échelle, en recherche appliquée pour découvrir de nouvelles molécules antiviralesmolécules antivirales ciblant d'autres virus, ou en recherche fondamentale, simplement pour mieux comprendre le fonctionnement du virus dans la cellule.