Jusqu'à récemment, le grand livre de l'information génétique semblait écrit dans un langage simple : quatre lettres (G, A, TT, C) pour l'ADN, la moléculemolécule qui stocke notre information génétique, et quatre (G, A, U, C) pour l'ARN, qui sert de support pour la traduction des protéines. Ce serait en fait plus compliqué, depuis que l'épigénétique est passée par là : elle montre que des modifications de l'ADN, comme des méthylations des lettres (ou nucléotides), jouent un rôle dans la régulation de l'expression des gènesgènes ; mais le nombre de nucléotides modifiés dans l'ARNARN est dix fois plus important que dans l'ADN ! Ces modifications permettent aux ARN, qu'il s'agisse d'ARNmARNm, ARNtARNt, ARN mitochondrial, etc., d'accomplir différentes fonctions.
Depuis quelque temps, il est admis que l'expression des gènes est également contrôlée par des modifications de l'ARN. Ainsi, la méthylation d'une adénosineadénosine en position 6 (m6A) affecte la localisation, la stabilité, la traduction et l'épissageépissage des ARNm. Cette modification spécifique de certaines régions de l'ARNm peut être lue par certaines protéines. Elle est dynamique et répond à des stimuli de l'environnement. Une enzymeenzyme (FTO) peut retirer le marquage m6A de l'ARNm ; cette méthylation est donc réversibleréversible. Ces découvertes récentes ont permis de créer un nouveau champ d'étude : l'épigénétique de l'ARN, qui étudie l'« épitranscriptome ».
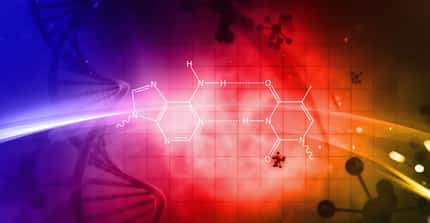
Cette fois, dans un nouvel article paru dans Nature, des chercheurs de l'université de Tel Aviv, du Sheba Medical Center et de l'université de Chicago, ont trouvé que l'ARN compte souvent une nouvelle lettre en plus : cette nouvelle modification de l'ARNm, correspond à une méthylation en position 1 de l'adénosine (m1A).
L’ARNm est traduit en protéine grâce au ribosome. © Designua, Shutterstock
Une nouvelle lettre clé pour le contrôle de l’expression du gène
La modification m1A a été localisée à une position proche du début de la traduction (près du codoncodon start), liée à une augmentation de la synthèse protéique. Présente chez les eucaryoteseucaryotes, des levureslevures aux mammifèresmammifères, cette modification aurait une proportion de l'ordre de 20 % chez les humains.
Cette forte conservation de la souris aux humains indique un rôle important joué par cette méthylation pour favoriser la traduction de l'ARNm méthylé. Comme l'explique Gidi Rechavi, chef du Centre de recherche sur le cancercancer au Sheba Medical Center, « ces nouvelles découvertes donnent à l'ARN une position centrale dans l'épigénétique ».
Ces travaux apportent de nouvelles informations sur le fonctionnement de l'ARN mais aussi son rôle dans le développement de maladies : « Nous nous attendons à ce qu'une perturbation de ce nouveau mécanisme de régulation soit associée à des états pathologiquespathologiques tels que le cancer et les maladies neurodégénérativesmaladies neurodégénératives ».
Les chercheurs étudient actuellement les processus cellulaires impliqués dans la lecture et l'effacement des m1A, ainsi que les voies biochimiques qui contrôlent cette nouvelle modification de l'ARN. Dans l'avenir, ils ont en projet d'explorer le rôle de cette méthylation dans le développement embryonnaire et son implication dans le cancer et les maladies neurodégénératives.