au sommaire
Les mécanismes de ces régulations sont maintenant bien connus. C'est en étudiant la régulation des enzymes permettant à E. coliE. coli de consommer le lactoselactose que Monod et Jacob ont pu proposer en 1961, le modèle dit de « l'opéron lactose », prototype de tous les modèles de régulation.
E. coli et l'opéron lactose
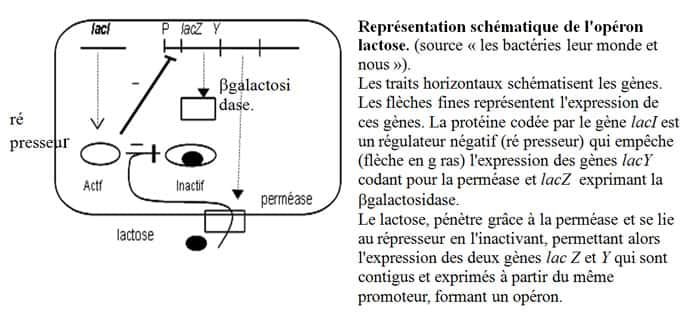
Pour utiliser le lactose comme source de carbone et d'énergie, E. coli dispose de deux protéinesprotéines spécifiques, une perméase membranaire qui fait pénétrer ce sucresucre dans son cytoplasmecytoplasme et une enzyme la β-galactosidase, qui transforme le lactose en glucoseglucose et galactose (qui sont métabolisés directement). En absence de lactose dans le milieu, les gènes codant ces protéines ne sont pas exprimés, parce qu'une protéine régulatrice est fixée sur l'ADNADN au niveau de la région où se trouvent ces deux gènesgènes et en empêche l'expression (le processus qui mène du gène à la protéine qu'il « encode »).
Lorsqu'une culture de ces bactériesbactéries ne dispose que de lactose comme source de carbone et d'énergie, cette petite molécule, si elle est en concentration suffisante, pénètre un peu dans les bactéries par diffusiondiffusion, et se lie à la protéine régulatrice en la modifiant de telle sorte qu'elle se détache de l'ADN, où les deux gènes précédemment réprimés peuvent être exprimés. C'est l'induction de la β-galactosidase par le lactose, qui est donc à la fois un nutrimentnutriment et un inducteur du système.
Ce modèle se décline sous de multiples variantes qu'il n'est pas question de recenser ici. Ce qui est constant c'est que certaines modifications de l'extérieur (composition chimique ou paramètre physiquephysique) sont perçues, car elles modifient la structure d'une ou plusieurs protéines relai, (souvent localisées dans la membrane des bactéries) ce qui entraîne (par des mécanismes divers, mais connus à présent) une modification au niveau de l'ADN qui permet ou empêche respectivement l'expression de gènes utiles ou inutiles à la vie dans les nouvelles conditions.
Protéines régulatrices et protéines réceptrices du signal
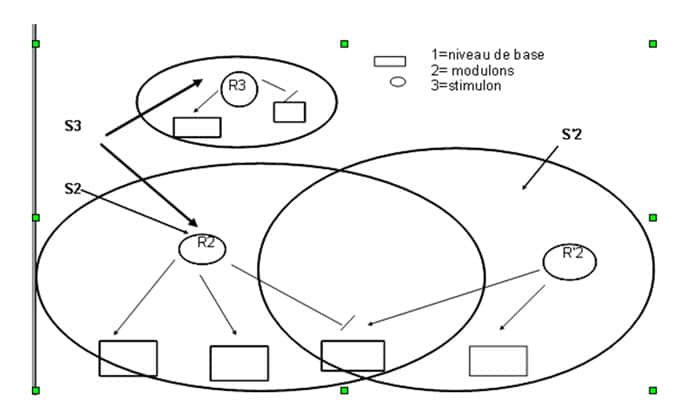
Les très nombreuses études consacrées à ces régulations se sont essentiellement focalisées dans deux directions. L'étude des protéines impliquées dans ces régulations : celles qui sont modifiées par la modification du milieu, (le « signal »), que l'on appelle sensors ou récepteurs du signal, les autres, dites régulatrices, qui reconnaissent des séquences d'ADN et permettent ou empêchent l'expression des gènes « cibles ». Et l'étude des interactions entre protéines et ADN et notamment les séquences de nucléotidesnucléotides et les structures protéiques qui permettent la reconnaissance protéine-ADN. Ce faisant, on a découvert que les protéines régulatrices ou sensors appartiennent à des « familles » trouvées dans de très nombreuses espècesespèces parfois très éloignées, et que les régulations forment des réseaux, avec des systèmes de plus en plus vastes et intégrés au fur et à mesure que les « signaux » modifient d'avantage de gènes, (donc de fonctions).
Bactéries et inductions
Mais ces réseaux, contrairement à ce que pourrait suggérer le schéma ci-dessus, ne sont pas statiques. Des observations concernant la dynamique de tels réseaux ont été rapportées au début de l'étude de l'opéron lactose, mais oubliées pendant près de quarante ans. Elles commencent tout juste à être redécouvertes et comprises. Des chercheurs avaient montré que, dans un milieu contenant de faibles concentrations d'inducteur, l'induction n'avait pas lieu. Cependant, si les bactéries avaient été soumises, ne serait-ce que 10 minutes, à une induction par une forte concentration d'inducteur avant d'être cultivées en présence d'une faible concentration, elles restaient induites pendant au moins 150 générations. Autrement dit, on avait une mémoire héritable du bref passage dans une forte concentration en lactose. Cette expérience avait deux types d'explication.
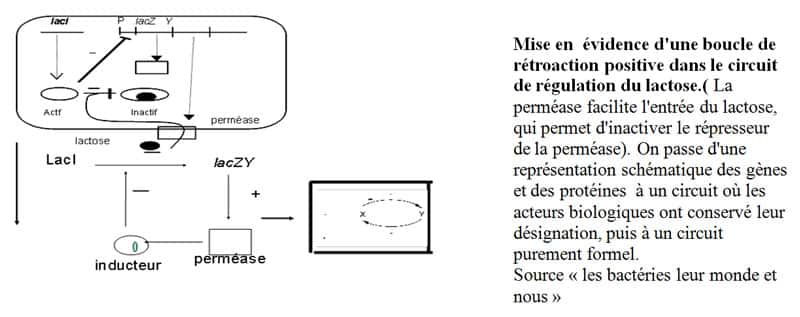
D'un point de vue particulier, la présence pendant 10 minutes d'une forte concentration d'inducteur dans le milieu avait permis d'induire la perméase grâce à laquelle, lorsque la concentration externe était faible, cette molécule pouvait tout de même pénétrer à l'intérieur des bactéries, et induire la synthèse des deux protéines. Au cours des générations suivantes, puisque, en se divisant par scissiparité, les bactéries conservent la moitié de la membrane antérieure, les perméases membranaires restent présentes et perpétuent la capacité à concentrer l'inducteur, capacité acquise pendant le séjour de 10 minutes en présence de forte concentration d'inducteur. La perméase est continuellement produite grâce à sa propre activité : c'est une boucle de rétroactionboucle de rétroaction positive.
La multistationnarité des bactéries : une modification épigénétique
D'un point de vue général, cette expérience met en évidence un type de régulation, pour lequel deux phénotypes différents (ici, induit ou non induit) peuvent se perpétuer pendant un très grand nombre de générations, dans des conditions identiques et sans qu'une mutation ne soit responsable de la différence. On appelle cela une modification épigénétique, qui correspond, dans ce cas, à un phénomène bien connu des physiciensphysiciens sous le nom de multistationnaritémultistationnarité. Il s'agit d'un processus qui fait partie des dynamiques des systèmes non linéaires, qu'il est bien sûr exclu de décrire ici, mais pour lequel une loi générale a pu être mise en évidence : la condition nécessaire pour qu'il y ait multistationnarité est qu'il existe une boucle de rétroaction positive dans le système de régulation. Ce qui est bien illustré par l'exemple décrit ici.
Conclusion sur les interactions des bactéries
Cet exemple n'est pas un cas isolé. Depuis quelques années, de multiples comportements dynamiques de ce type ont été découverts chez les bactéries, (et chez les autres organismes vivants). Mais les comportements dynamiques en biologie sont généralement « non linéaires » et nécessitent alors l'usage de modélisationmodélisation mathématique (ou de simulation informatiquesimulation informatique) pour être compris. Et la biologie n'est pas, pour le moment, armée pour utiliser ces méthodes, qui requièrent des collaborations avec des mathématiciensmathématiciens ou des bio-informaticiensbio-informaticiens. Il reste encore beaucoup à découvrir.
De plus, dans cet exemple, comme dans la majorité de ceux étudiés jusqu'à présent, les modifications analysées concernent les comportements dynamiques des bactéries elles-mêmes. Si on étend l'étude aux transformations, non moins dynamiques des environnements, on verra que s'ouvre un vaste champ de compréhension des adaptations bactéries/environnements pour le moment encore largement inexploré.
Ainsi, puisque les bactéries ne se comprennent que dans leurs interactions avec leurs environnements, et que ces interactions ne sont pas statiques, mais se modifient constamment selon des dynamiques diverses, le plus souvent non linéaires, il faut se donner les moyens d'intégrer les études dynamiques en microbiologie (comme dans toute la biologie d'ailleurs), ce qui étend fortement la gamme des méthodes ou des collaborations nécessaires.